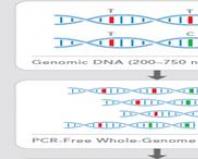
DNA-Chips. Supramolekularer Nanodruck. Verwendung in der Medizin und Genetik
Fertigungstechnologie und Design sind die Hauptdeterminanten für die Vor- und Nachteile biologischer Mikrochips, ihre Anwendungen, Preismerkmale und Gesamtverfügbarkeit.
Zur Herstellung von DNA-Chips gibt es zwei grundsätzlich unterschiedliche Ansätze: die Synthese von DNA einer vorgegebenen Sequenz direkt auf einer Matrix und die chemische Immobilisierung vorsynthetisierter Oligonukleotide auf einem Substrat. Die Technologie zur Synthese von Oligonukleotiden auf einem Substrat mittels Photolithographie ist patentiert und wird von Affymetrix verwendet, einem weltweit führenden Hersteller von DNA-Chips, der bis zu 70 % des Weltmarktes kontrolliert. Die Technologie basiert auf der Verwendung einer photolabilen Schutzgruppe für monomere DNA-Einheiten, die durch Bestrahlung mit UV-Licht vom terminalen Rest des auf dem Substrat synthetisierten Oligonukleotids entfernt wird. Der Vorteil dieser Technologie ist die Möglichkeit, Chips mit einer sehr hohen Abscheidungsdichte herzustellen – bis zu 100.000 Punkte pro 1 cm2. Der offensichtliche Nachteil der Methode ist die Komplexität und die hohen Kosten des Prozesses.
Technologien zur chemischen Immobilisierung von DNA-Fragmenten auf festen Substraten wurden vor etwa 30 Jahren entwickelt und werden derzeit weiter verbessert. Das allgemeine Prinzip der Immobilisierung biologischer Moleküle besteht in der Bildung eines Paares chemischer Gruppen auf der Oberfläche des Substrats und am Ende des zusammengefügten Oligonukleotids, wodurch die Bildung einer kovalenten Bindung zwischen ihnen sichergestellt wird. Существует огромное количество таких способов, большинство из которых основано на взаимодействии нуклеофильной группы (например, аминогруппы), находящейся на поверхности подложки или привязанной к молекуле олигонуклеотида, с электрофильным агентом, в роли которого могут выступать тем или иным путем активированные карбоновые кислоты, моноэфиры фосфорной кислоты usw.
Beurteilung der Genexpression mithilfe von DNA-Microarrays ( am Beispiel von Affymetrix GeneChip)
Die häufigste Verwendung von DNA-Microarrays ist die Beurteilung der Genexpression. Die beliebteste Plattform zur Lösung dieser Art von Problemen sind Affymetrix GeneChip-Microarrays, die kurze Oligonukleotidsequenzen verwenden, um in einer RNA-Probe enthaltene Gene zu identifizieren. Das Vorhandensein jedes Gens in der Probe wird mithilfe eines Satzes von Sonden mit einer Länge von jeweils 25 Nukleotiden nachgewiesen. Um die Qualität des Experiments zu verbessern, werden für jede betrachtete Sequenz mehrere Kopien von Sonden auf dem Chip platziert.
Affymetrix-Mikroarrays verwenden typischerweise 11 bis 20 Probenpaare pro interessierendem Gen. Eine Komponente solcher Paare, eine so genannte Perfect-Match-Sonde (PM), ist genau komplementär zur Sequenz des entsprechenden Gens – man geht davon aus, dass es seine RNA ist, die sich an die PM-Sonde bindet. Diese Verbindung wird als spezifische Hybridisierung bezeichnet. Es können jedoch auch Nukleotidsequenzen anderer Gene an die Sonden angehängt werden (unspezifische Hybridisierung). Um die Auswirkungen der unspezifischen Hybridisierung zu beurteilen, werden die anderen Komponenten des Paares verwendet – Sonden, die als Mismatch-Sonde (MM) bezeichnet werden. Die darin enthaltene Nukleotidsequenz stimmt mit der Sequenz in den entsprechenden PM-Sonden überein, wobei das zentrale (dreizehnte) Nukleotid durch ein komplementäres ersetzt wurde. Das Verhältnis der Lumineszenzintensität von PM- und MM-Proben wurde ursprünglich verwendet, um den Effekt der unspezifischen Hybridisierung zu neutralisieren, spätere Studien stellten jedoch die Richtigkeit dieses Ansatzes in Frage.
Genotypisierung des gesamten Genoms polymorpher Loci mithilfe von Mikroarrays mit hoher Dichte ( am Beispiel des Illumina Human610-Quad BeadChip)
Bisher haben führende Hersteller (Illumina, Affymetrix, Sequenom usw.) Plattformen mit hochdichten Microarrays für die Genotypisierung und Genexpressionsanalyse entwickelt (Abb. 30).
Illumina Human610-Quad BeadChip umfasst mehr als 600.000 Einzelnukleotidpolymorphismen und Marker der Genkopienzahlvariation (CNV). Jeder SNP für den Biochip wird auf der Grundlage genauer Informationen ausgewählt, um die Effizienz bei der Bestimmung des Zusammenhangs mit der Krankheit zu verbessern. Es gibt mehr als 10 Millionen SNPs im menschlichen Genom und die Untersuchung jedes polymorphen Locus im Genom ist wirtschaftlich nicht machbar. Illumina hat einen einzigartigen, rationalen Ansatz zur SNP-Auswahl entwickelt, der hochwertige Genotypisierungsdaten und die Abdeckung aller notwendigen Loci für die Analyse der Krankheitsanfälligkeit gewährleistet.
Abb.30. Chip-Technologien für die Hochdurchsatz-Genotypisierung
Illumina-Biochips umfassen eine Reihe von SNPs, sogenannte Marker- oder Tag-SNPs, die als Proxy-Marker (repräsentative Marker) für alle gängigen SNPs (mit einer seltenen Allelhäufigkeit ≥ 5 %) im Genom verwendet werden können. Die Wahl der Marker-SNPs basiert auf dem Ausmaß des Bindungsungleichgewichts (r 2) zwischen nahe beieinander liegenden polymorphen Loci. Der hohe r2-Wert zwischen zwei SNPs, der auf eine hohe Korrelation hinweist, macht diese SNPs zu guten Proxy-Markern. Bei einem maximalen r 2 von 1 befinden sich die beiden SNPs in einem vollständigen Bindungsungleichgewicht und können als absolute Proxy-Marker dienen, d. h. Sie müssen einen SNP genotypisieren, um den Genotyp eines anderen herauszufinden (The Power of Intelligent SNP Selection (www.illumina.com)). Die Verwendung von Marker-SNPs ermöglicht es, die maximale Informationsmenge, ein hohes Maß an Genomabdeckung und Genabdeckung zu erhalten und außerdem die Größe der Studienstichprobe zu reduzieren, ohne die Effizienz der Bestimmung genetischer Assoziationen zu beeinträchtigen. Die genomische Abdeckung ist definiert als die Anzahl der SNPs, die sich in einem Bindungsungleichgewicht mit einem Referenzsatz von Loci befinden. Illumina-Spezialisten verwenden alle im HapMap-Projekt genotypisierten SNPs als Referenzsatz. Laut Illumina bietet der Human610-Quad BeadChip-Biochip eine Abdeckung von 89 % des Genoms in europäischen Populationen (CEU, HapMap), 86 % des Genoms in asiatischen (CHB+JPT, HapMap) und 58 % des Genoms in afrikanischen ( YRI, HapMap) ( bei r 2 > 0,8). Der durchschnittliche Abstand zwischen den Markern auf diesem Biochip beträgt 4,7 kb, der Median liegt bei 2,7 kb.
Die BeadArray-Technologie von Illumina basiert auf der Verwendung von 3-Mikron-Silizium-Mikrokügelchen (Beads), die sich entweder auf Bündeln optischer Fasern oder auf flachen Siliziumwafern zu Mikrovertiefungen zusammenfügen. Jede Mikrosphäre ist mit Tausenden Kopien spezifischer Oligonukleotide bedeckt, die ortsspezifische und Adresssequenzen enthalten, wobei letztere bestimmen, welches Kügelchen in welche Mikrozelle eingebettet ist.
Im ersten Schritt der Genotypisierung erfolgt die genomweite Amplifikation von DNA-Proben, anschließend erfolgt die Fragmentierung der DNA (Abb. 31). Im nächsten Schritt werden unmarkierte Fragmente von DNA-Proben mit den entsprechenden 50-Nukleotid-Sonden hybridisiert, die auf den Mikrosphären des Chips fixiert sind. Anschließend wird zur genauen Identifizierung des Allels mithilfe des Enzyms DNA-Polymerase ein markiertes komplementäres Nukleotid an das Testnukleotid angehängt. Der zweistufige Nachweis von Allelen für jeden Marker – Hybridisierung mit 50-Nukleotid-Sonden und anschließender enzymatischer Nachweis des Nukleotids – sorgt für eine hohe Selektivität und Spezifität für die Allelidentifizierung (Infinium® HD DNA Analysis BeadChips (www.illumina.com)).


Reis. 31. Schema des Genotypisierungsprotokolls unter Verwendung von Illumina Human610 quad BeadChip-Biochips (Infinium® HD DNA Analysis BeadChips (www.illumina.com).
Daher diskutiert das vorgestellte Lehrbuch die wichtigsten molekulargenetischen Methoden zur Untersuchung erblicher Erkrankungen des Menschen. In einem Buch ist es unmöglich, alle derzeit existierenden Forschungsmethoden zu beschreiben oder molekulardiagnostische Protokolle für jede spezifische Form der erblichen Pathologie vorzustellen. Dieses Lehrbuch vermittelt einen allgemeinen Überblick über die Methodik und Strategie der molekulargenetischen Diagnostik und zeigt die Methoden auf, die in der Praxis eines modernen Forschers am häufigsten verwendet werden. Die vorgeschlagenen Forschungsprotokolle, eine detaillierte Beschreibung verschiedener Methoden mit Abbildungen, sollen Studierenden der medizinischen und biologischen Ausbildung mit Schwerpunkt Genetik dabei helfen, molekulargenetische Methoden zur Analyse des menschlichen Genoms erfolgreich zu beherrschen.
LISTE DER VERWENDETEN REFERENZEN
1. Bochkov N.P. Klinische Genetik. M. 2011. – 592 S.
2. Glick B., Pasternak J. Molekulare Biotechnologie. Prinzipien und Anwendung. - Moskau: Mir, 2002. - 589 S.
3. Gorbunova V. N., Baranov V. S. Einführung in die molekulare Diagnostik und Gentherapie erblicher Erkrankungen. Sankt Petersburg. „Sonderliteratur“. 1997. – 287 S.
4. Davis K. Genomanalyse. Methoden. M. 1990. – 246 S.
5. Eprintsev A.T., Popov V.N., Fedorin D.N. Identifizierung und Untersuchung der Genexpression. // Pädagogisches und methodisches Handbuch für Universitäten. - Woronesch. – 2008. – 63 S.
6. Schimulew I.F. Allgemeine und molekulare Genetik. N. 2003. – 479 S.
7. Inge-Vechtomov S.G. Genetik mit den Grundlagen der Selektion. M. - 2010. – 718 S.
8. Kuzmina N.A. Grundlagen der Biotechnologie, 2005 // http://www.biotechnolog.ru/
9. Kulmamabetova G. Probenvorbereitung. Methoden zur DNA/RNA-Isolierung // Moderne Probleme der Biologie, ENU, Astana, 2012, Vorlesung 9.
10. Menschikova V.V. Klinische und laboranalytische Technologien und Geräte. M. 2007. – 240 S.
11. Molekulare Biotechnologie. Prinzipien und Anwendung / hrsg. N.K. Jankowski. – M.: Mir, 2002. – 588 S.
12. Molekulare klinische Diagnostik. Methoden / Hrsg. S. Herrington, J. McGee. – M.: Mir, 1999. – 558 S.
13. Mutovin G.R. Klinische Genetik. Genomik und Proteomik der nicht investigativen Pathologie: ein Lehrbuch. M. 2010. – 832 S.
14. Mukhacheva T.A., Kovalev S.Yu. Angewandte Bioinformatik. Sonderkurs. UrFU, Jekaterinburg. 2012.
15. Patrushev L.I. Genexpression. M. Naturwissenschaften. 2000. – 830 S.
16. Polyanichko A. M. Elektrophorese in PAAG. Toolkit. Sankt Petersburg. 2007.
17. Pritchard D.D. – Visuelle medizinische Genetik. – pro. aus dem Englischen bearbeitet von N.P. Botschkowa. – M.: GEOTAR-Media. – 2009. – 200 S.
18. Puzyrev V.P., Stepanov V.A. Pathologische Anatomie des menschlichen Genoms. N. – 1997. – 224 S.
19. Echtzeit-PCR / Hrsg. D.V. Rebrikova. - M.: Binom., 2009. – 223 S.
20. Severin E.S. Biochemie: Lehrbuch. M. 2004. – 784 S.
21. Sukachev M. Moderne Methoden der gesamten Genomsequenzierung (Dekodierung) von DNA bei der Diagnose und Behandlung von Krankheiten // http://innoros.ru/publications/articles/13/
22. Telkov M. Echtzeit-PCR: methodische Grundlagen, Optimierung, Anwendung. Bio-Red-Labors.
23. Theoretische Grundlagen der Polymerase-Kettenreaktion. NPO „DNA-Technology“ – Moskau, 1998 // www.dna-technology.ru
24. Shatz V.D., Sakhartova O.V. Hochleistungsflüssigkeitschromatographie. Grundlagen der Theorie. Methodik. Anwendung in der medizinischen Chemie.
25. Mathew C.C. Die Isolierung hochmolekularer eukaryotischer DNA // Methoden in der Molekularbiologie / ED. Walker J.M. – N.Y.: Haman-Presse. – 1984. – S. 31–34.
26. Nolan T, Hands RE, Bustin SA (2006). „Quantifizierung von mRNA mittels Echtzeit-RT-PCR.“ Nat. Protokoll. 1:1559–1582. DOI:10.1038/nprot.2006.236. PMID 17406449.
27. VanGuilder HD, Vrana KE, Freeman WM (2008). „25 Jahre quantitative PCR zur Genexpressionsanalyse.“ Biotechniken 44: 619–626. DOI:10.2144/000112776. PMID 18474036.
ANWENDUNG
Anhang 1.
Erstellung von Richtlinien zur DNA-Extraktion
Verwandte Informationen.
DNA-Mikrochip(dt. DNA-Microarray) ist eine komplexe Technologie, die in der Molekularbiologie und Medizin eingesetzt wird. Ein DNA-Mikrochip ist eine kleine Oberfläche, auf der Fragmente einzelsträngiger synthetischer DNA mit bekannter Sequenz in hoher Dichte und in einer bestimmten Reihenfolge aufgetragen sind. Diese Fragmente fungieren als Sonden, mit denen komplementäre DNA-Stränge aus der untersuchten Probe, die normalerweise mit einem Fluoreszenzfarbstoff markiert sind, hybridisieren (doppelsträngige Moleküle bilden). Je mehr DNA-Moleküle mit einer bestimmten Sequenz in der Probe vorhanden sind, desto mehr von ihnen binden an die komplementäre Sonde und desto stärker ist das optische Signal an der Stelle auf dem Mikrochip, an der die entsprechende Sonde „eingepflanzt“ wurde. Nach der Hybridisierung wird die Oberfläche des Mikrochips gescannt, und als Ergebnis wird jeder DNA-Sequenz ein bestimmter Signalpegel zugewiesen, der proportional zur Anzahl der in der Mischung vorhandenen DNA-Moleküle mit einer bestimmten Sequenz ist.
In einem herkömmlichen DNA-Mikrochip (wie etwa von Affymetrix) sind die Sonden auf einer festen Oberfläche befestigt – einem Glas- oder Silikonchip. Andere Plattformen, beispielsweise die von Illumina, verwenden mikroskopisch kleine Perlen anstelle großer fester Oberflächen. Die DNA-Microarray-Technologie findet in der modernen Biologie und Medizin vielfältige Anwendungsmöglichkeiten zur Analyse komplexer DNA-Mischungen – beispielsweise der Gesamtheit aller Transkripte (Messenger-RNA) in einer Zelle. DNA-Microarrays werden zur Analyse von Veränderungen in der Genexpression, zur Identifizierung einzelner Nukleotidpolymorphismen, zur Genotypisierung oder zur Neusequenzierung mutierter Genome verwendet. Mikrochips unterscheiden sich in Design, Betriebsfunktionen, Genauigkeit, Effizienz und Kosten.
Ein Beispiel für die Verwendung eines DNA-Microarrays
Nachfolgend finden Sie ein Beispiel für ein Experiment mit einem DNA-Microarray.

- Biologische Proben werden zum Vergleich isoliert oder gezüchtet. Sie können sich auf dieselben Individuen vor und nach einer Behandlung beziehen (bei paarweisen Vergleichen) oder auf verschiedene Gruppen von Individuen, zum Beispiel Kranke und Gesunde usw.
- Gereinigte Nukleinsäure wird aus der Probe isoliert und ist Gegenstand der Untersuchung: Dies kann RNA in einer Genexpressionsprofilierungsstudie, DNA in einer vergleichenden genomischen Hybridisierungsstudie usw. sein. Dieses Beispiel entspricht dem ersten Fall.
- Die Qualität und Quantität der gewonnenen Nukleinsäure wird überprüft. Sind die Voraussetzungen erfüllt, kann das Experiment fortgesetzt werden.
- Basierend auf den verfügbaren RNA-Proben werden durch den Prozess der reversen Transkription komplementäre DNA-Sequenzen (cDNA) synthetisiert.
- Während des Prozesses der Amplifikation (Synthese zusätzlicher DNA-Kopien) erhöht sich die Anzahl der cDNA-Sequenzen in den Proben um ein Vielfaches.
- An den Enden der cDNA-Sequenzen werden fluoreszierende oder radioaktive Markierungen angebracht.
- Die resultierenden Proben, gemischt mit den notwendigen Chemikalien, werden durch ein mikroskopisches Loch auf DNA-Mikrochips aufgetragen und der Hybridisierungsprozess beginnt, bei dem eine der cDNA-Ketten an die auf dem Mikrochip vorhandene Komplementärkette gebunden wird.
- Nachdem der Hybridisierungsprozess abgeschlossen ist, werden die Chips gewaschen, um jegliches restliches Material zu entfernen.
- Die resultierenden Mikrochips werden mit einem Laser gescannt. Das Ergebnis sind ein- oder zweifarbige Bilder (abhängig von der Anzahl der verwendeten Farbstoffe).
- Jedem Bild wird ein Gitter überlagert, sodass jede seiner Zellen einem Abschnitt des Chips mit Proben desselben Typs entspricht. Der Leuchtintensität von Proben in einer Gitterzelle wird eine bestimmte Zahl zugeordnet, die in allererster Näherung als Maß für die Anzahl der in der entsprechenden Probe vorhandenen RNA-Sequenzen dienen kann.
Die weitere Verarbeitung der Ergebnisse erfordert eine mehrstufige Einbindung komplexer statistischer Apparate.
Vorverarbeitung experimenteller Daten
Die Korrelation zwischen den Intensitäten zweier Proben aus demselben DNA-Mikroarray, die dasselbe Gen repräsentieren, beträgt typischerweise mehr als 95 %. Diese Tatsache wird oft als Bestätigung der guten Reproduzierbarkeit von Experimenten mit Chips interpretiert. Wenn jedoch dasselbe biologische Material in zwei Teile geteilt wird und daraus unterschiedliche Mikroarrays hergestellt werden, liegt die Korrelation zwischen den resultierenden Intensitäten höchstwahrscheinlich zwischen 60 und 80 %. Die Korrelation zwischen Chips und Proben von Mäusen aus demselben Wurf kann bis zu 30 % betragen. Wenn Experimente in verschiedenen Laboren durchgeführt werden, kann die Korrelation zwischen ihren Ergebnissen noch geringer sein.
Diese geringe Reproduzierbarkeit der Intensitäten ist auf die kombinierten Effekte einer großen Anzahl von Variationsquellen zurückzuführen. Sie lassen sich in drei große Gruppen einteilen. Die biologische Variation umfasst die inhärenten Eigenschaften von Organismen. Technische Abweichungen treten in der Phase der Probenisolierung, -färbung und -hybridisierung auf. Der Messfehler ist mit dem Scannen fertiger Arrays verbunden, deren Ergebnisse beispielsweise durch Staub im Inneren des Scanners beeinträchtigt werden können.
Die Neutralisierung der Auswirkungen technischer Variationen und Messfehler erfolgt in der Phase der Vorverarbeitung von DNA-Mikroarrays.
Hintergrundkorrektur
Die Notwendigkeit einer Hintergrundkorrektur ist mit dem Vorhandensein von Störfaktoren wie Rauschen des optischen Erkennungssystems (die beim Scannen erhaltenen Intensitätsdaten stimmen nicht mit den „echten“ Probenintensitäten überein) und unspezifischer Hybridisierung (Anheftung von Nukleotidsequenzen an fremde Sonden) verbunden Proben).
Normalisierung
Durch die Datennormalisierung ist es möglich, mehrere in einem Experiment betrachtete Chips miteinander vergleichbar zu machen. Das Hauptziel der Analyse in dieser Phase besteht darin, den Einfluss systematischer nichtbiologischer Unterschiede zwischen Mikroarrays auszuschließen. Die Ursachen für solche Unterschiede sind vielfältig: Unterschiede in der Effizienz der reversen Transkription, Farbstoffmarkierung, Hybridisierung, physikalische Unterschiede zwischen Chips, geringfügige Unterschiede in den Reagenzkonzentrationen und Unterschiede in den Laborbedingungen.
Es zeigt sich, dass die Wahl der Normalisierungsmethode einen erheblichen Einfluss auf das Ergebnis der Analyse hat.
Zusammenfassung
Verallgemeinerung der Expressionsniveauwerte für alle Proben, die denselben Sequenzen entsprechen
Qualitätskontrolle
Emissionsverarbeitung
Die Hauptphase der statistischen Verarbeitung
Links
- DNA-Mikroarray
- DNA-Microarray-Experiment – Artikel aus der englischen Wikipedia
- DNA Microarray Virtual Lab – interaktives Schritt-für-Schritt-Beispiel für ein zweifarbiges DNA-Microarray-Experiment
- Zehn Fallstricke der Microarray-Analyse – häufige Fehler bei der DNA-Microarray-Analyse
Spezifische Reihenfolge. Ein Oligonukleotid kann ein kurzer Abschnitt eines Gens oder einer anderen DNA-Komponente sein, der zur Hybridisierung mit cDNA oder mRNA verwendet wird. Die Sonden-Ziel-Hybridisierung wird mithilfe von Fluoreszenz oder Chemilumineszenz nachgewiesen und quantifiziert, wodurch die relative Menge an Nukleinsäure einer bestimmten Sequenz in einer Probe bestimmt werden kann.
Bei einem herkömmlichen DNA-Mikroarray werden Sonden kovalent an eine feste Oberfläche – einen Glas- oder Siliziumchip – gebunden. Andere Plattformen, beispielsweise die von Illumina, verwenden mikroskopisch kleine Perlen anstelle großer fester Oberflächen. DNA-Mikroarrays unterscheiden sich von anderen Mikroarrays nur dadurch, dass sie zur Messung von DNA oder als Teil eines komplexeren DNA-Erkennungs- und Analysesystems verwendet werden.
DNA-Microarrays werden zur Analyse von Veränderungen in der Genexpression, zur Identifizierung einzelner Nukleotidpolymorphismen, zur Genotypisierung oder zur Neusequenzierung mutierter Genome verwendet. Mikrochips unterscheiden sich in Design, Betriebsfunktionen, Genauigkeit, Effizienz und Kosten.
Geschichte
Anmerkungen
Wikimedia-Stiftung. 2010.
Sehen Sie in anderen Wörterbüchern, was ein „DNA-Mikrochip“ ist:
Der Begriff DNA-Microarray Der Begriff im Englischen DNA-Microarray Synonyme DNA-Chip, DNA-Chip, Genchip, DNA-Chip Abkürzungen Verwandte Begriffe Biosensor, Genom, DNA, DNA-Sonde, Labor auf einem Chip, RNA, Oligonukleotid Definition Miniaturplatte mit... . ..
Begriff DNA-Sonde Begriff im Englischen DNA-Sonde Synonyme Abkürzungen Verwandte Begriffe biologische Nanoobjekte, biomedizinische mikroelektromechanische Systeme, Biosensor, Genom, DNA, DNA-Mikrochip, Labor auf einem Chip, Oligonukleotid... ... Enzyklopädisches Wörterbuch der Nanotechnologie
Begriff DNA Begriff im Englischen DNA Synonyme Desoxyribonukleinsäure Abkürzungen DNA Verwandte Begriffe Genabgabe, Aktuator, Bakteriophage, Proteine, biologische Nanoobjekte, Biomimetik, biomimetische Nanomaterialien, Gentechnik,... ... Enzyklopädisches Wörterbuch der Nanotechnologie
DNA-Chip
DNA-Biochip- DNA-Chip DNA-Chip (auch: DNA-Biochip, DNA-Mikrochip, DNA-Nanochip) Ein spezieller Chip zur Erkennung genetischer Mutationen oder Verschiebungen und zur Diagnose von Krankheiten. Ein Biochip für die amerikanische Armee, entwickelt von Spezialisten aus... ... Erklärendes englisch-russisches Wörterbuch zur Nanotechnologie. - M.
Mikrochip-Gen-M-Microarray- Mikrochip, Gen-Chip oder Mikrochip * Microarray oder Gen-Chip oder Mikrochip, ein Satz Tausender einzigartiger bekannter einzelsträngiger DNA-Fragmente, die auf einer festen Basis immobilisiert sind. Diese Fragmente repräsentieren alles... ... Genetik. Enzyklopädisches Wörterbuch
Sir Edwin Mallor Southern (* 7. Juni 1938), englischer Molekularbiologe, Mitglied der Royal Society for the Advancement of Natural Knowledge (auch bekannt als Royal Society of London), Träger des Lasker-Preises (2005). Der Preis war... Wikipedia
DNA-Mikroarray mit komplementärer DNA. Komplementäre DNA (cDNA) ist DNA, die aus reifer mRNA in einer durch Reverse Transkriptase katalysierten Reaktion synthetisiert wird. Cha cDNA ... Wikipedia
Die quantitative Nukleinsäureanalyse bestimmt die Konzentration von DNA oder RNA in einer Mischung oder einem reinen Präparat. Reaktionen mit Nukleinsäuren erfordern häufig genaue Informationen über die Menge und Reinheit des Arzneimittels. Um die Konzentration zu bestimmen... ... Wikipedia
Der Begriff Amplifikation Der Begriff im Englischen Amplifikation Synonyme Abkürzungen Verwandte Begriffe Definition (lat. amplificatio Verstärkung, Steigerung), in der Molekularbiologie eine Erhöhung der Anzahl von DNA-Kopien. Beschreibung In einer Zelle erfolgt die Verstärkung in... ... Enzyklopädisches Wörterbuch der Nanotechnologie
Genexpression ist ein Prozess, bei dem Erbinformationen eines Gens (DNA-Nukleotidsequenz) in ein funktionelles Produkt – RNA oder Protein – umgewandelt werden. Die Regulierung der Genexpression ermöglicht es Zellen, ihre eigene Struktur und Funktion zu kontrollieren und ist die Grundlage für Zelldifferenzierung, Morphogenese und Anpassung.
DNA-Chips sind ein einzigartiges Analysewerkzeug, mit dem Sie das Vorhandensein bestimmter DNA-Sequenzen in einer analysierten Probe (normalerweise biologischen Ursprungs) bestimmen können (sog. Hybridisierungsanalyse). Die Analyse mittels DNA-Chips ist um ein Vielfaches günstiger als der Einsatz alternativer Technologien (Elektrophorese, Echtzeit-PCR) und ermöglicht mit einem einfachen Detektor die Arbeit außerhalb des Labors.
Zum ersten Mal DNA-Chips wurden Ende der 1980er Jahre in der Forschung eingesetzt. Diese mittlerweile weit verbreitete Methode, die eine gleichzeitige Analyse der Expression vieler Gene ermöglicht, basiert auf dem Prinzip der Erkennung von mRNA- oder cDNA-Zielen durch deren Hybridisierung mit einzelsträngigen DNA-Fragmenten, die auf einem Mikrochip immobilisiert sind. Ein moderner DNA-Mikrochip besteht aus Tausende von Desoxyoligonukleotiden (Sonden oder Proben), die in Form mikroskopischer Punkte gruppiert und auf einem festen Träger fixiert sind. Jeder Punkt enthält mehrere Pikomol DNA mit einer spezifischen Nukleotidsequenz. DNA-Microarray-Oligonukleotide können kurze Abschnitte von Genen oder anderen funktionellen DNA-Elementen sein und werden zur Hybridisierung mit cDNA oder mRNA (cRNA) verwendet. Die Sonden-Ziel-Hybridisierung wird mithilfe von Fluoreszenz oder Chemilumineszenz nachgewiesen und quantifiziert, wodurch die relative Menge an Nukleinsäure einer bestimmten Sequenz in einer Probe bestimmt werden kann.
Bei einem herkömmlichen DNA-Mikroarray werden Sonden kovalent an eine feste Oberfläche – einen Glas- oder Siliziumchip – gebunden. Andere Plattformen, beispielsweise die von Illumina, verwenden mikroskopisch kleine Perlen anstelle großer fester Oberflächen.
DNA-Microarrays werden zur Analyse von Veränderungen in der Genexpression, zur Identifizierung einzelner Nukleotidpolymorphismen, zur Genotypisierung oder zur Neusequenzierung mutierter Genome verwendet. Mikrochips unterscheiden sich in Design, Betriebsfunktionen, Genauigkeit, Effizienz und Kosten.
DNA-Microarrays:
cDNA-Microarrays
Oligonukleotid
(zweifarbig mit Fluoreszenzerkennung)
Oligonukleotid
(Affymetrix, monochromatisch mit Fluoreszenzdetektion)
Membran-c-DNA-Mikroarrays
(mit radioaktiver Detektion)
Gel-c-DNA-Chips
Protein-Mikrochips
Eine kleine Geschichte
1980er: Proteinchips
~1991: Chemie der DNA-Synthese auf einem Substrat (hohe Dichte) – Affymetrix-Oligonukleotid-Chips (Fodor, Stryer, Lockhart)
~1995: Mikrograbungsroboter – cDNA-Chips der Stanford University (Pat Brown und Dari Shalon)
1990er Jahre: MPI-Gelchips
Allerdings schlugen Augenlicht und Kobrin bereits 1982 ein DNA-Array vor ( Krebs Forschung) und stellten 1984 einen Chip mit 4000 Elementen für die Untersuchung von Krebszellen her.
(Der Artikel wurde abgelehnt WissenschaftUnd Natur)
Was kann mit DNA-Microarrays untersucht werden?
Genexpression in verschiedenen Geweben
Genexpression in normalen und pathologischen Zuständen (in normalen und Krebszellen)
Veränderungen der Genexpression im Laufe der Zeit durch äußere Einflüsse (Zellinteraktion mit einem Krankheitserreger, Medikament)
Ausdrucksprofile(Muster) unterscheiden sich zwischen normalen und Krebszellen oder verschiedenen Krebsarten. Heilbare und unheilbare Arten von Leukämie erzeugen unterschiedliche Muster. Anhand der Art der Muster ist es möglich, den Krankheitsverlauf bereits in einem sehr frühen Stadium mit hoher Wahrscheinlichkeit vorherzusagen.
Expressions-Mikrochips
Einer der aktiv entwickelten Bereiche der Microarray-Technologie ist die Untersuchung von Transkriptionsprofilen bei komplexen Krankheiten. Obwohl alle Zellen in unserem Körper die gleiche vererbte genomische DNA besitzen, exprimiert jede Zelle je nach Zelltyp, biologischen Prozessen, normalem oder pathologischem Zustand usw. unterschiedliche Gene in Form von mRNA. Diese Vielfalt der Genexpressionsprofile wurde aufgrund ihrer biologischen und klinischen Bedeutung intensiv untersucht. Die Fähigkeit der Microarray-Technologie, die Expression von Hunderten und Tausenden von Genen zu analysieren, hat sich bei der Entschlüsselung einer so komplexen Krankheit wie Krebs als besonders gefragt erwiesen. Die Microarray-Technologie ermöglicht es, die Expression von Zehntausenden Genen gleichzeitig zu überwachen und so ein molekulares Porträt einer Zelle zu erstellen. Zu den wichtigsten Auswirkungen der Genexpressionsprofilierung gehören Diagnose, Stratifizierung und Prognose bei vielen Krebsarten. Obwohl die histopathologische Beurteilung, ergänzt durch zytogenetische Untersuchung und Analyse mehrerer molekularer Marker, immer noch der Goldstandard für die Diagnosestellung und Prognosebestimmung ist, deuten neuere Arbeiten darauf hin, dass sie in vielen Fällen durch Genexpressionsprofile ersetzt werden kann. Diagnose und Prognose von Krebs erfordern die gemeinsame Expertise mehrerer Behandler, wie Onkologen, Pathologen und Zytogenetiker, zudem können die endgültigen Schlussfolgerungen je nach methodischen Ansätzen und Qualifikation der Experten variieren. Mikrochips könnten die Bemühungen vieler Spezialisten vollständig ersetzen, darüber hinaus die Genauigkeit von Diagnose und Prognose erhöhen und eine einzige standardisierte Plattform für die Analyse bereitstellen.
Zur Analyse der Genexpression werden zwei Arten von Microarrays verwendet: basierend auf komplementärer DNA (cDNA) und basierend auf Oligonukleotidsonden. Bei cDNA-basierten Mikroarrays handelt es sich um DNA-Fragmente, die auf der Oberfläche von Standard-Mikroskopgläsern oder anderen festen Trägern angebracht sind. In Oligonukleotid-Mikroarrays werden Oligonukleotide mit einer Länge von 25–60 Nukleotidbasen (nt) auf demselben Substrat immobilisiert. Das Probenvorbereitungsverfahren für die Microarray-Analyse ist in Abb. dargestellt. 2. Gesamt-RNA wird aus Zellen isoliert (manchmal wird auch eine mRNA-Fraktion isoliert), dann wird eine Reverse-Transkriptionsreaktion unter Verwendung eines kombinierten Primers durchgeführt, der eine zum polyA-terminalen Fragment der mRNA komplementäre Sequenz und eine Region der T7-RNA-Polymerase enthält Promoter. Der Einschluss der T7-RNA-Polymerase-Promotorsequenz in die synthetisierte cDNA-Kette ermöglicht weitere In-vitro-Amplifikationsreaktionen: Das T7-RNA-Polymerase-Enzym produziert viele Kopien der RNA von jedem cDNA-Molekül in einem Reagenzglas. Auf diese Weise erfolgt eine lineare Amplifikation der ursprünglichen mRNA. In der Regel werden die resultierenden RNA-Moleküle gleichzeitig markiert, indem bei der Reaktion Nukleotide mit einer Fluoreszenzmarkierung verwendet werden. Bei Experimenten mit Oligonukleotid-Mikroarrays wird häufig eine Art fluoreszierender Markierung verwendet, um eine Probe komplementärer RNA (cRNA) zu markieren, und die Genexpressionsniveaus werden durch Vergleich der resultierenden Fluoreszenzsignale mit denen der internen Kontrollpunkte des Mikroarrays bestimmt. Beim Arbeiten mit cDNA-basierten Microarrays werden im Experiment in der Regel zwei Proben verwendet: Die Kontrollprobe wird mit einem Fluoreszenzfarbstoff markiert, die Testprobe mit einem anderen, dann werden sie gemischt und mit einem Mikrochip hybridisiert. Basierend auf dem Verhältnis zweier verschiedener Fluoreszenzmarkierungen in jeder Zelle des Mikrochips wird eine Zunahme oder Abnahme des Expressionsgrads eines bestimmten Gens beurteilt. Unabhängig von der Technologieplattform generiert jedes Experiment Daten, die eine Schätzung des Expressionsgrads von Zehntausenden und Hunderttausenden Genen enthalten. Um eine solche Datenmenge zu verarbeiten, wird ein ziemlich komplexer mathematischer Apparat verwendet, in erster Linie die Clusteranalyse. Microarray-Daten können in Bezug auf klinische Daten (hypothesengesteuerte Analyse) oder ohne Rücksicht auf klinische Merkmale des Patienten (unüberwachte Analyse) analysiert werden.
Klassische Methoden ermöglichen die gleichzeitige Analyse der Expression mehrerer Gene oder erfordern beispielsweise den Einsatz spezialisierter Microarray-Technologien wie z Affymetrix. Affymetrix verwendet eine Kombination aus Photolithographie und chemischer Oligonukleotidsynthese, um GeneChip®-Mikroarrays herzustellen.
GELB- Wenn das Gen sowohl in erkranktem (Cy5) als auch in normalem (Cy3) Gewebe exprimiert wird, hybridisiert DNA, die sowohl mit roten als auch mit grünen Farbstoffen markiert ist, an dieser Stelle, was zu einer gelben Farbe führt
ROT- Wenn das Gen nur in erkranktem (Cy5) Gewebe exprimiert wird, hybridisiert an dieser Stelle nur mit rotem Farbstoff markierte DNA
GRÜN- Wenn das Gen nur in gesundem (Cy3) Gewebe exprimiert wird, hybridisiert an dieser Stelle nur mit grünem Farbstoff markierte DNA
SCHWARZ– wenn das Gen weder in erkranktem noch in gesundem Gewebe exprimiert wird
Auf diese Weise,
DNA-Microarrays ermöglichen die gleichzeitige Analyse von Informationen über die Expression vieler tausend Gene.
Die derzeit hauptsächlich verwendeten Arten von DNA-Microarrays sind cDNA-Microarrays und Oligonukleotid-Chips von Affymetrix.
cDNA-Mikroarrays basieren auf der Hybridisierung gemischter Versuchs- und Kontrollproben, die mit verschiedenen Fluoreszenzfarbstoffen markiert sind, auf einem Chip, auf dessen Oberfläche doppelsträngige c-DNA aufgebracht ist, die etwa 10.000–20.000 Genen entspricht.
Affimetrix-Mikrochips basieren auf der Hybridisierung von Biotin-markierter cRNA einer experimentellen Probe mit einem Satz von Perfect Match- und Mismatch-Oligonukleotiden, die auf einem Chipsubstrat synthetisiert wurden, gefolgt von einer Färbung mit Streptavidin-Phycoerythrin. GeneChip Human Genome U133 Plus 2.0 ermöglicht die gleichzeitige Analyse von 47.000 Transkripten, darunter 38.500 charakterisierte Gene. Der Mikrochip umfasst 1.300.000 Oligonukleotide unterschiedlicher Art.
Die Analyse der gewonnenen Daten erfordert eine mehrstufige mathematische Verarbeitung mit speziellen statistischen Methoden.
Praktisch gesehen ermöglicht der Einsatz von Mikrochips heute die Lösung folgender Probleme:
genaue Diagnose, Identifizierung neuer Subtypen der Krankheit, Klärung der Klassifikation;
Vorhersage des Krankheitsverlaufs und des klinischen Ergebnisses, Identifizierung von Genen und Signalwegen, die an der Pathogenese onkohämatologischer Erkrankungen beteiligt sind, Suche nach neuen Angriffspunkten für eine gezielte differenzierte Therapie;
Entwicklung und Schaffung einfacherer und kostengünstigerer Diagnosetests, einschließlich solcher, die auf der Mikrochip-Technologie basieren (Mikrochips, die Proben für Dutzende oder Hunderte von Genen anstelle von Zehntausenden und Hunderttausenden enthalten);
Einbeziehung von Microarrays in prospektive klinische Studien, Bestätigung der Ergebnisse der Microarray-Analyse zur Aufnahme in klinische Behandlungsprotokolle, Gestaltung klinischer Protokolle unter Berücksichtigung neuer Daten über die Art von Krankheiten, die mithilfe der Microarray-Technologie gewonnen wurden.
DNA-Microarrays
DNA-Chips sind ein einzigartiges Analysewerkzeug, mit dem Sie das Vorhandensein bestimmter DNA-Sequenzen in einer analysierten Probe (normalerweise biologischen Ursprungs) bestimmen können (sog. Hybridisierungsanalyse). Die Analyse mittels DNA-Chips ist um ein Vielfaches günstiger als der Einsatz alternativer Technologien (Elektrophorese, Echtzeit-PCR) und ermöglicht mit einem einfachen Detektor die Arbeit außerhalb des Labors.
DNA-Chips wurden erstmals Ende der 80er Jahre des letzten Jahrhunderts in der Forschung eingesetzt. Diese mittlerweile weit verbreitete Methode, die eine gleichzeitige Analyse der Expression vieler Gene ermöglicht, basiert auf dem Prinzip der Erkennung von mRNA- oder cDNA-Zielen durch deren Hybridisierung mit einzelsträngigen DNA-Fragmenten, die auf einem Mikrochip immobilisiert sind.
Ein DNA-Chip ist ein fester Träger, auf dem einzelsträngige DNA-Fragmente unterschiedlicher Länge (normalerweise kovalent) immobilisiert sind: kurz – 15–25 Nukleotide, lang – 25–60 Nukleotide und cDNA-Fragmente – von 100 bis 3000 Nukleotide. Als Substratmaterialien kommen Glas, Silizium, verschiedene Polymere, Hydrogele (zum Beispiel auf Basis von Polyacrylamid) und sogar Gold zum Einsatz. Die häufigsten Substrate sind Glas.
Protein- und Peptidchips
Zur Analyse der Produkte der Gentranslation werden Chips verwendet, die auf der Basis von Polypeptiden aufgebaut sind. Die meisten Wirkstoffziele sind Proteine, daher können Protein- und Peptidchips für die Wirkstoffentwicklung nützlich sein. Protein-Mikrochips können in der Medizin als Miniatur-Analysesysteme zur Bestimmung des Immunstatus des Körpers, zur Erkennung allergischer Sensibilisierung und zur Identifizierung spezifischer Allergene äußerst nützlich sein. Mikrochips, die eine Sammlung der Hauptantigene der wichtigsten Krankheitserreger (Bakterien, Pilze und Viren) darstellen, ermöglichen es, Blutproben gleichzeitig auf das Vorhandensein von Hunderten und Tausenden von Antikörpern zu analysieren und Infektionen schnell zu identifizieren. Signalaufzeichnungsmethoden sind von große Bedeutung bei der Entwicklung von Protein-Mikrochips. Dazu gehören: die allererste bekannte Methode – RIA (Radioimmunoassay), bei der eine radioaktive Markierung verwendet wird, Immunoassay mit fluoreszierenden Markierungen – FIA und Enzyme-Linked Immunosorbent Assay (ELISA), bei dem die Markierung ein Enzymmolekül ist, das kovalent an ein Antikörpermolekül gebunden ist . Als Marker im ELISA werden hochaktive stabile Enzyme (alkalische Phosphatase, Peroxidase usw.) ausgewählt. Der Vorteil von ELISA ist die Möglichkeit der mehrfachen Signalverstärkung. In den letzten Jahren wurden empfindliche Substratsysteme entwickelt, die unlösliche fluoreszierende Produkte wie ELF-97 liefern. Offensichtlich muss der Prozess der Herstellung eines Protein-Mikrochips den Vorgang der Fixierung und Immobilisierung auf dem Mikrochip umfassen. Die Wahl der Methode wird von vielen Parametern bestimmt – der Beschaffenheit des Ausgangssubstrats, dem späteren Einsatzgebiet des Mikrochips usw. Protein-Mikrochips werden vor allem für die Analyse aller bekannten (und verfügbaren) biologischen Flüssigkeiten aktiv eingesetzt, darunter Blutserum/-plasma, Urin, Liquor, Speichel, Tränenflüssigkeit, Fruchtwasser usw.